【中国发明,中国发明授权】基于能量和概率的局部结构胃癌耐药lncRNA二级结构预测方法
有权-审定授权 中国
- 申请号:
- CN201910897339.9
- 专利权人:
- 吉林大学
- 授权公告日/公开日:
- 2021.03.05
- 专利有效期:
- 2019.09.23-2039.09.23
- 技术分类:
- 转化方式:
- 转让
- 价值度指数:
-
- 55.0分
- 价格:
- 面议
- 专利信息&法律状态
- 专利自评
- 专利技术文档
- 价值度指数
- 发明人阵容
 著录项
著录项
- 申请号
- CN201910897339.9
- 申请日
- 20190923
- 公开/公告号
- CN110648719A
- 公开/公告日
- 20200103
- 申请/专利权人
- [吉林大学]
- 发明/设计人
- [王林宇, 刘元宁, 钟晓丹, 郑少阁, 张浩, 董立岩, 朱晓冬, 刘海明]
- 主分类号
- G16B15/30
- IPC分类号
- C12N 9/0008(2013.01) C12N 9/16
- CPC分类号
- C12N 9/0008(2013.01) C12N 9/16(2013.01)
- 分案申请地址
- 国省代码
- 吉林(22)
- 颁证日
- G06T1/00
- 代理人
- [鞠传龙]
 摘要
摘要
本发明公开了一种基于能量和概率的局部结构胃癌耐药lncRNA二级结构预测方法,其方法为:步骤一、将数据读到缓存单元中;步骤二、得到其二级结构预测结果;步骤三、获得一组长度为50的短RNA序列集合M;步骤四、得到序列中所有的局部次优结构集;步骤五、找最优的局部结构组件;步骤六、得到当前的lncRNA二级结构预测结果;步骤七、集合交给最优结构获取单元;步骤八、通过RS‑232串口传回到上位机的显示单元上进行输出显示。有益效果:该方法可以查找出具有特定局部结构的功能组件,分析和解释lncRNA的功能,最后交由生物实验进行功能验证,为高效筛选胃癌耐药相关lncRNA提供一条新的思路。
 法律状态
法律状态
| 法律状态公告日 | 20210305 |
| 法律状态 | 授权 |
| 法律状态信息 | 授权 |
| 法律状态公告日 | 20210305 |
| 法律状态 | 授权 |
| 法律状态信息 | 授权 |
| 法律状态公告日 | 20200204 |
| 法律状态 | 实质审查的生效 |
| 法律状态信息 | 实质审查的生效 IPC(主分类):G16B 15/30 专利申请号:2019108973399 申请日:20190923 |
| 法律状态公告日 | 20200103 |
| 法律状态 | 公开 |
| 法律状态信息 | 公开 |
 权利要求
权利要求
权利要求数量(1)
独立权利要求数量(1)
1.一种基于能量和概率的局部结构胃癌耐药lncRNA二级结构预测方法,其特征在于:其方法如下所述:
步骤一、通过上位机的输入单元输入lncRNA序列,胃癌耐药蛋白及其对应mRNA信息,并通过RS-232串口传至lncRNA二级结构预测装置的内存储单元,并进一步将数据读到缓存单元中;
步骤二、预处理单元从缓存单元中读取胃癌耐药蛋白及其对应mRNA信息,分析胃癌耐药蛋白对应的mRNA 5’端UTR区、3’端UTR区的结构特性,用概率模型对这两个UTR区进行结构预测,得到其二级结构预测结果,并将统计结果中的茎环特征存于Stru5、Stru3中;
步骤三、局部次优单元从缓存单元中读出lncRNA序列,记录序列长度L,利用长度为50的动态移动窗口从LncRNA的5’端开始截取lncRNA序列,动态移动窗口的步长大小设置为1,当动态移动窗口的末端到达LncRNA的3’端时,移动停止,此时共获得一组长度为50的短RNA序列集合M;
步骤四、针对M中的每一条短RNA序列,用概率模型得到序列中所有的局部次优结构集,具体过程如下:
i)从集合中的第一条短RNA序列开始,读取集合中的一条序列,通过概率模型得到序列中所有可能的茎区集合,其中可能包含假结茎区;
ii)设置相对最小能量和最小能量变化阈值的能量模型,根据该能量模型寻找出当前茎区集合内的所有相容的茎区集合,并拼接其中的相容茎区,得到基于当前窗口内RNA序列的包含假结次优局部结构集;
步骤五、在局部最优单元中,从预处理单元中读取茎环结构特征Stru5、Stru3,在次优局部结构集中找最优的局部结构组件,具体如下:
i)如果集合不为空,则从中随机取一个次优局部结构;
ii)将Stru5、Stru3与选取次优局部结构进行结构比对,判断是否能与Stru5、Stru3匹配或互补,如能则将该次优局部结构存储为LocM[i],否则转iii;
iii)如没有找到与Stru5、Stru3匹配或互补的次优局部结构,则按波尔兹曼分布采样得到次优局部结构并存储为LocM[i];
iv)删除中的其它次优局部结构;
v)从M中删除该短RNA序列;
步骤六、从M中重新选取一条短RNA序列,重复进行步骤四和步骤五两步操作,直至集合M为空,从内存储单元中取出所有局部最优结构进行组装拼接,得到当前的lncRNA二级结构预测结果;
步骤七、将动态移动窗口的长度加5,重复进行步骤三、步骤四、步骤五和步骤六四步操作,直至窗口的长度为200;此时就可以得到当前所有的lncRNA二级结构预测结果集合,其中,将该集合交给最优结构获取单元;
步骤八、在最优结构获取单元中,根据能量模型和概率模型,计算集合中每个lncRNA结构的最小自由能和概率,获取自由能能量小且概率模型大的结构,作为为当前全局最优的lncRNA二级结构;将得到的全局最优lncRNA二级结构存于外存储单元中,并可通过RS-232串口传回到上位机的显示单元上进行输出显示。
1.一种基于能量和概率的局部结构胃癌耐药lncRNA二级结构预测方法,其特征在于:其方法如下所述:
步骤一、通过上位机的输入单元输入lncRNA序列,胃癌耐药蛋白及其对应mRNA信息,并通过RS-232串口传至lncRNA二级结构预测装置的内存储单元,并进一步将数据读到缓存单元中;
步骤二、预处理单元从缓存单元中读取胃癌耐药蛋白及其对应mRNA信息,分析胃癌耐药蛋白对应的mRNA 5’端UTR区、3’端UTR区的结构特性,用概率模型对这两个UTR区进行结构预测,得到其二级结构预测结果,并将统计结果中的茎环特征存于Stru5、Stru3中;
步骤三、局部次优单元从缓存单元中读出lncRNA序列,记录序列长度L,利用长度为50的动态移动窗口从LncRNA的5’端开始截取lncRNA序列,动态移动窗口的步长大小设置为1,当动态移动窗口的末端到达LncRNA的3’端时,移动停止,此时共获得一组长度为50的短RNA序列集合M;
步骤四、针对M中的每一条短RNA序列,用概率模型得到序列中所有的局部次优结构集,具体过程如下:
i)从集合中的第一条短RNA序列开始,读取集合中的一条序列,通过概率模型得到序列中所有可能的茎区集合,其中可能包含假结茎区;
ii)设置相对最小能量和最小能量变化阈值的能量模型,根据该能量模型寻找出当前茎区集合内的所有相容的茎区集合,并拼接其中的相容茎区,得到基于当前窗口内RNA序列的包含假结次优局部结构集;
步骤五、在局部最优单元中,从预处理单元中读取茎环结构特征Stru5、Stru3,在次优局部结构集中找最优的局部结构组件,具体如下:
i)如果集合不为空,则从中随机取一个次优局部结构;
ii)将Stru5、Stru3与选取次优局部结构进行结构比对,判断是否能与Stru5、Stru3匹配或互补,如能则将该次优局部结构存储为LocM[i],否则转iii;
iii)如没有找到与Stru5、Stru3匹配或互补的次优局部结构,则按波尔兹曼分布采样得到次优局部结构并存储为LocM[i];
iv)删除中的其它次优局部结构;
v)从M中删除该短RNA序列;
步骤六、从M中重新选取一条短RNA序列,重复进行步骤四和步骤五两步操作,直至集合M为空,从内存储单元中取出所有局部最优结构进行组装拼接,得到当前的lncRNA二级结构预测结果;
步骤七、将动态移动窗口的长度加5,重复进行步骤三、步骤四、步骤五和步骤六四步操作,直至窗口的长度为200;此时就可以得到当前所有的lncRNA二级结构预测结果集合,其中,将该集合交给最优结构获取单元;
步骤八、在最优结构获取单元中,根据能量模型和概率模型,计算集合中每个lncRNA结构的最小自由能和概率,获取自由能能量小且概率模型大的结构,作为为当前全局最优的lncRNA二级结构;将得到的全局最优lncRNA二级结构存于外存储单元中,并可通过RS-232串口传回到上位机的显示单元上进行输出显示。
 说明书
说明书
技术领域
本发明涉及一种局部结构胃癌耐药lncRNA二级结构预测方法,特别涉及一种基于能量和概率的局部结构胃癌耐药lncRNA二级结构预测方法。
背景技术
目前,胃癌多药耐药的发生是涉及多个蛋白编码基因、多条信号通路的复杂调控网络共同作用的结果,针对上述任何耐药相关分子的单一干预难以有效逆转胃癌多药耐药。
长链非编码RNA(long non-coding RNA,lncRNA)是一类内源性、单链、非编码的大分子RNA。近年来的大量研究表明lncRNA虽然不能被翻译成蛋白质,但它对许多重要的生命活动在多个层次上进行调控,其功能涉及基因转录调控、翻译后修饰和表观遗传学控制等多个方面。
通过对胃癌耐药相关分子进行初步研究,发现有多条在胃癌耐药细胞系中差异表达显著的lncRNA可能调控胃癌耐药蛋白PrPc表达。由此推断,lncRNA可能通过调控耐药蛋白的表达,在胃癌耐药中起着重要的作用,靶向干预lncRNA,可能更有效地逆转胃癌耐药。
胃癌耐药细胞中时空特异表达lncRNA数量众多,单纯依靠传统生物学实验采取逐条敲除、抑制或过表达的手段在细胞系中处理候选lncRNA,对于每一个实验对照组都可能需要测定质谱或制作转录组相关分子的表达谱芯片,甚至还要建立动物模型进行在体实验。这个流程在研究对象规模较大时无论从时间还是人力、物力成本的角度考量,都不具有良好的可操作性。生物信息学可通过合理的数学建模、利用强大的计算资源,保证在研究成功的前提下,从海量的信息中挖掘出研究人员所关切的、有研究价值的数据,合理的缩小研究对象范围。
目前预测lncRNA二级结构的方法存在以下几种问题:
1)实验手段预测lncRNA二级结构存在的问题
实验上常采用X光造影或核磁共振技术获得RNA二级结构,但长链RNA在体外实验环境中极易降解,很难结晶;采用降解了RNA的不饱和环区,通过拼接子结构来推测RNA分子二级结构,破坏了可能发挥生物功能的环区,影响了结构准确性;SHAPE技术分析了RNA骨架任意位置的单链灵活性,推测碱基是否配对,但无法确定配对对象。
由此可见,采用实验手段,仍无法获得准确的lncRNA结构信息,需要借助计算机来进行lncRNA结构预测。
2)现有计算机方法预测lncRNA二级结构的缺陷
现有的预测方法可分为两种:一种基于多条序列,典型的是比较分析法,基于已存在的结构模板,在多条同源序列中寻找公共结构,但lncRNA缺乏结构模板,无法进行比较分析;另一种是单条RNA序列的预测方法,基于实验测定的短序列结构(十几个碱基长)的自由能,利用最近邻能量模型寻找全局结构,但最近邻能量模型存在缺陷,且该种方法只对较短序列有效,序列长度增加,计算方法花费巨大,精度大幅降低。
计算机方法普遍存在两个问题:第一是只关注全局结构的准确性,忽略了可能发挥生物功能的局部结构;第二是随着序列长度的增长,计算花费和存储消耗增加,精度大幅降低。上述方法都不适用于lncRNA的结构预测问题。
发明内容
本发明的目的是为了解决目前预测lncRNA二级结构的方法中所存在的诸多问题而提供的一种基于能量和概率的局部结构胃癌耐药lncRNA二级结构预测方法。
本发明提供的基于能量和概率的局部结构胃癌耐药lncRNA二级结构预测方法,其方法如下所述:
步骤一、通过上位机的输入单元输入lncRNA序列,胃癌耐药蛋白及其对应mRNA信息,并通过RS-232串口传至lncRNA二级结构预测装置的内存储单元,并进一步将数据读到缓存单元中;
步骤二、预处理单元从缓存单元中读取胃癌耐药蛋白及其对应mRNA信息,分析胃癌耐药蛋白对应的mRNA 5’端UTR区、3’端UTR区的结构特性,用概率模型对这两个UTR区进行结构预测,得到其二级结构预测结果,并将统计结果中的茎环特征存于Stru5、Stru3中;
步骤三、局部次优单元从缓存单元中读出lncRNA序列,记录序列长度L,利用长度为50的动态移动窗口从LncRNA的5’端开始截取lncRNA序列,动态移动窗口的步长大小设置为1,当动态移动窗口的末端到达LncRNA的3’端时,移动停止,此时共获得一组长度为50的短RNA序列集合M;
步骤四、针对M中的每一条短RNA序列,用概率模型得到序列中所有的局部次优结构集,具体过程如下:
i)从集合中的第一条短RNA序列开始,读取集合中的一条序列,通过概率模型得到序列中所有可能的茎区集合,其中可能包含假结茎区;
ii)设置相对最小能量和最小能量变化阈值的能量模型,根据该能量模型寻找出当前茎区集合内的所有相容的茎区集合,并拼接其中的相容茎区,得到基于当前窗口内RNA序列的包含假结次优局部结构集;
步骤五、在局部最优单元中,从预处理单元中读取茎环结构特征Stru5、Stru3,在次优局部结构集中找最优的局部结构组件,具体如下:
i)如果集合不为空,则从中随机取一个次优局部结构;
ii)将Stru5、Stru3与选取次优局部结构进行结构比对,判断是否能与Stru5、Stru3匹配或互补,如能则将该次优局部结构存储为LocM[i],否则转iii;
iii)如没有找到与Stru5、Stru3匹配或互补的次优局部结构,则按波尔兹曼分布采样得到次优局部结构并存储为LocM[i];
iv)删除其它次优局部结构;
v)从M中删除该短RNA序列;
步骤六、从M中重新选取一条短RNA序列,重复进行步骤四和步骤五两步操作,直至集合M为空,从内存储单元中取出所有局部最优结构进行组装拼接,得到当前的lncRNA二级结构预测结果;
步骤七、将动态移动窗口的长度加5,重复进行步骤三、步骤四、步骤五和步骤六四步操作,直至窗口的长度为200;此时就可以得到当前所有的lncRNA二级结构预测结果集合,其中,将该集合交给最优结构获取单元;
步骤八、在最优结构获取单元中,根据能量模型和概率模型,计算集合中每个lncRNA结构的最小自由能和概率,获取自由能能量小且概率模型大的结构,作为当前全局最优的lncRNA二级结构;将得到的全局最优lncRNA二级结构存于外存储单元中,并可通过RS-232串口传回到上位机的显示单元上进行输出显示。
上述方法中所用装置是由上位机、ARM9微处理器的接口部分、存储单元及处理单元构成。
上位机由输入单元、接口部分的USB接口、JTAG调试接口、以太网接口、RS-232串口和显示单元共同构成来完成与ARM9微处理器的协调工作;其中输入单元与接口部分012连接,负责完成RNA序列的输入;接口部分负责与ARM9微处理器进行连接通信;显示单元与接口部分012连接,负责完成lncRNA预测结果的输出显示。
ARM9微处理器的接口部分包括:USB接口、JTAG调试接口、以太网接口及RS-232串口;其中USB接口可与U盘连接,实现将二级结构识别得到的结果数据的转存,以此实现存储单元的扩增;JTAG调试接口,通过JTAG仿真(编程器)转换设备与上位机JTAG接口相连,用来实现程序的在线调试;以太网接口通过此接口与上位机的以太网接口进行连接,从而实现ARM9微处理器与上位机的互通信;RS-232串口通过此接口与上位机的RS-232串口进行连接,从而实现ARM9微处理器与上位机的互通信。
存储单元包括内存储单元、缓存单元及外存储单元,内存储单元与缓存单元进行连接,负责完成lncRNA初始数据及中间数据的存储;缓存单元与内存储单元及预处理单元进行连接,负责完成lncRNA二级结构预测的初始数据的存储;外存储单元与最优结构获取单元进行连接,负责完成lncRNA二级结构预测的结果数据的存储。
处理单元包括预处理单元、局部次优单元、局部最优结构单元及最优结构获取单元,预处理单元与缓存单元连接,负责获取缓存单元中lncRNA数据读入及mRNA分析处理;局部次优单元负责初始的局部组件序列,并实现局部组件序列的截取及局部组件的滑窗调整和移动,利用ACRNA算法得到所有局部次优结构集;局部最优单元找到最优的局部结构,存于内存储单元中;最优结构获取单元对于内存储单元0222中存储的所有局部最优结构进行比较,最终获取出全局最优结构。
本发明的有益效果:
本发明提供的基于能量和概率的局部结构胃癌耐药lncRNA二级结构预测方法能够从lncRNA结构组件的角度阐述胃癌耐药现象以及如何逆转这一现象的客观规律。基于实验获得的候选胃癌耐药相关lncRNA,通过局部结构融合的lncRNA结构预测模型预测其二级结构,有效的利用了lncRNA的局部结构的稳定性;将预测精度较低的长序列转化为多个预测精度高的短序列结构的组合可以提高预测准确度且降低了预测的资源消耗;该方法可以查找出具有特定局部结构的功能组件,分析和解释lncRNA的功能,最后交由生物实验进行功能验证,为高效筛选胃癌耐药相关lncRNA提供一条新的思路。
附图说明
图1为本发明所述方法中所用系统结构原理示意图。
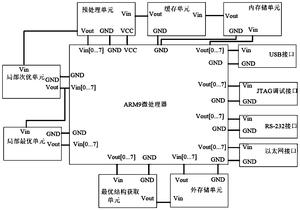
图2为本发明所用系统电路原理示意图。
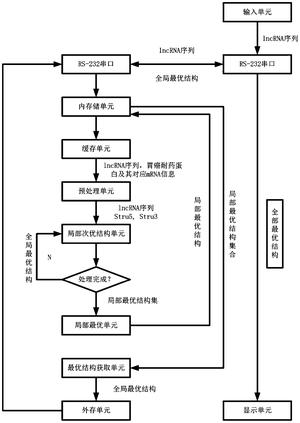
图3为本发明所述方法逻辑功能示意图。
具体实施方式
请参阅图1至图3所示:
如图1所示,本发明的基于滑动窗口组件式胃癌耐药lncRNA二级结构的预测模型装置与上位机连接硬件结构图,包括上位机及ARM9微处理器构成的lncRNA二级结构预测装置。
在本发明的实施例中,通常利用一台通用的PC计算机作为上位机,该上位机可通过RS-232串口和基于三星公司生产的32位的ARM920T核的微处理器的lncRNA二级结构识别装置进行连接,共同作用以完成lncRNA二级结构的预测。
上位机的输入单元及显示单元均采用PC计算机的输入及输出设备来实现其功能。
本发明中可通过上位机的以太网接口及ARM9微处理器的以太网接口实现上位机与ARM9微处理器的互通信,以太网接口采用DM9000完全综合的、成本较低的单一快速以太网控制器芯片。
本发明中,增加了上位机的JTAG调试接口及ARM9微处理器的JTAG调试接口,将此类接口通过JTAG仿真(编程器)转换设备进行连接,可以实现上位机实时地对ARM9微处理器上程序的分析和执行的监控。
本发明中,USB接口采用的是USB2.0接口,为了实现存储单元的扩增,可以将lncRNA二级结构预测结果数据通过上位机的USB接口或ARM9微处理器的USB接口转存到U盘。
ARM9微处理器系统程序存储单元选用的是32M Hynix公司的HY57V561620CTSDRAM作为内存储单元,64M SAMSUNG公司的K9F1208UOM Nand Flash作为缓存单元,及1G的硬盘作为扩展外存储单元。
ARM9微处理器的处理单元中所包含的各单元均是固化在ARM9微处理器上的lncRNA二级结构算法,并且在运算时使用32位运算部件。
如图2所示,是基于滑动窗口组件式胃癌耐药lncRNA二级结构的预测装置电路原理图,其连接关系如下:USB接口、JTAG调试接口、以太网接口及RS-232串口的数据输入口Vin分别与ARM9微处理器的数据输出引脚Vout1[0..7]相连,其GND分别与ARM9微处理器的GND相连。
内存储单元的数据输入口Vin与ARM9微处理器的数据输出引脚Vout1[0..7]相连,其数据输出口Vout与缓存单元的数据输入口Vin相连,其GND与ARM9微处理器的GND相连。缓存单元的数据输入口Vin与内存储单元的数据输出口Vout相连,其数据输出口Vout与预处理单元的数据输入口Vin相连,其GND与ARM9微处理器的GND相连。外存储单元的数据输入口Vin与最优结构获取单元的数据输出口Vout相连,其数据输出口Vout与ARM9微处理器的数据输入引脚Vin1[0..7]相连,其GND与ARM9微处理器02的GND相连。
预处理单元的数据输入口Vin与缓存单元的数据输出口Vout相连,其数据输出口Vout与分别与ARM9微处理器的数据输入引脚Vin1[0..7]及局部次优单元的数据输入口Vin相连,其GND与ARM9微处理器的GND相连,局部次优单元的数据输入口Vin与预处理单元的数据输出口Vout相连,其数据输出口Vout分别与ARM9微处理器的数据输入引脚Vin1[0..7]及局部最优单元的数据输入口Vin相连,其GND与ARM9微处理器的GND相连。局部最优单元的数据输入口Vin与局部次优单元的数据输出口Vout相连,其数据输出口Vout与ARM9微处理器的数据输入引脚Vin1[0..7]相连,其GND与ARM9微处理器的GND相连。最优结构获取单元的数据输入口Vin与ARM9微处理器的数据输出引脚Vout1[0..7]相连,其数据输出口vout与外存储单元0201的数据输入口Vin相连,GND与ARM9微处理器的GND相连。
工作步骤:
如图3逻辑功能图所示,用户使用该装置进行lncRNA二级结构预测的步骤如下:
步骤一、通过上位机的输入单元输入lncRNA序列,胃癌耐药蛋白及其对应mRNA信息。并通过RS-232串口传至lncRNA二级结构预测装置的内存储单元,并进一步将数据读到缓存单元中;
步骤二、预处理单元从缓存单元中读取胃癌耐药蛋白及其对应mRNA信息,分析胃癌耐药蛋白对应的mRNA 5’端UTR区、3’端UTR区的结构特性,用ACRNA对这两个UTR区进行结构预测,统计其中的茎环特征存于Stru5、Stru3中;
步骤三、局部次优单元从缓存单元中读出lncRNA序列,并利用滑窗设定的大小截取组件序列,组件式滑窗尺寸的变化范围设为50nt-200nt,初始滑窗大小为50nt,将滑窗放于lncRNA序列的5’起始位置,滑窗计数器i=1。用概率模型和能量模型得到所有局部次优结构集LocS;
步骤四、在局部最优单元中,从预处理单元中读取Stru5、Stru3,在次优局部结构集LocS中找最优的局部结构组件LocM[i]。
步骤五、再次从缓存单元中读出序列信息,局部次优单元对组件滑窗进行移动,将滑窗尺寸加5,读取新的RNA子序列,转至步骤三重复操作,直至滑窗移到lncRNA序列的3’端;若对所有组件已处理完成,则从内存储单元中取出所有局部最优结构进行组装拼接,得到当前尺寸滑窗下的lncRNA集合lncStru[i],将滑窗尺寸加5,转至步骤三,直至滑窗尺寸为200nt;存储所有滑窗尺寸下预测得到的lncRNA二级结构lncStru[i],i=1…31;交给最优结构获取单元020805;
步骤六、在最优结构获取单元中,根据能量模型和概率模型,计算每个lncRNA结构的最小自由能和概率,获取自由能能量小且概率模型大的结构,为当前全局最优的lncRNA二级结构;将得到的全局最优lncRNA二级结构存于外存储单元中,并可通过RS-232串口传回到上位机的显示单元上进行输出显示。
为验证本专利的有效性,我们选取了ABBA01177720这条LncRNA来做验证,这条LncRNA的长度为1020nt,验证步骤如下:
步骤一、输入LBHZ02002032序列,胃癌耐药蛋白及其对应mRNA信息到内存储单元和缓存单元中;
步骤二、预处理单元读取出胃癌耐药蛋白及其对应mRNA序列信息,分析胃癌耐药蛋白对应的mRNA 5’端UTR区、3’端UTR区的结构特性,用概率模型对这两个UTR区进行结构预测,并将得到预测结果存于Stru5、Stru3中;
步骤三、局部次优单元从缓存单元中读出LBHZ02002032序列,记录序列长度L。使用长度为50、步长大小为1的动态移动窗口截取RNA序列片段,得到一个包含971个短的RNA序列的集合;
步骤四、针对集合中的每一条短RNA序列,用概率模型得到序列中所有可能的茎区集合,用能量模型拼接相容茎区,得到包含假结次优局部结构集。
步骤五、利用步骤二得到Stru5、Stru3在次优局部结构集中找该短序列的最优的局部结构组件。一个短序列的得到一个最优的局部结构组件。
步骤六、从集合中重新选取一条短RNA序列,重复进行步骤四和步骤五两步操作,直至集合内全部的短序列均操作完成,共得到971个局部最优结构。利用能量模型从内存储单元中取出所有局部最优结构进行组装拼接,得到当前窗口大小下的lncRNA二级结构预测结果。
步骤七、将动态移动窗口的长度加5,重复进行步骤三、步骤四、步骤五和步骤六四步操作,直至窗口的长度为200;此时我们可以得到31个不同窗口下的lncRNA二级结构预测结果
步骤八、针对上步得到的31个预测结果,利用能量模型和概率模型,计算每个结果中每个结果所对应的最小自由能和概率大小,获取自由能能量小且概率模型大的结构,作为为当前全局最优的lncRNA二级结构;
步骤九、将所得到的结果与其他预测二级结构的方法进行的对比,在预测的敏感性和特异性上均优于其他方法,证明本专利提供的方法是有效的。
 价值度评估
价值度评估
技术价值
经济价值
法律价值
0 0 055.0分
0 50 75 100专利价值度是通过科学的评估模
型对专利价值进行量化的结果,
基于专利大数据针对专利总体特
征指标利用计算机自动化技术对
待评估专利进行高效、智能化的
分析,从技术、经济和法律价值
三个层面构建专利价值评估体
系,可以有效提升专利价值评估
的质量和效率。
总评:55.0分
该专利价值中等 (仅供参考)
本专利文献中包含【1 个实施例】、【1 个技术分类】,从一定程度上而言上述指标的数值越大可以反映出所述专利的技术保护及应用范围越广。 【专利权的维持时间6 年】专利权的维持时间越长,其价值对于权利人而言越高。
技术价值 29.0
该指标主要从专利申请的著录信息、法律事件等内容中挖掘其技术价值,专利类型、独立权利要求数量、无效请求次数等内容均可反映出专利的技术性价值。 技术创新是专利申请的核心,若您需要进行技术借鉴或寻找可合作的项目,推荐您重点关注该指标。
部分指标包括:
授权周期(发明)
17 个月独立权利要求数量
1 个从属权利要求数量
0 个说明书页数
6 页实施例个数
1 个发明人数量
8 个被引用次数
0 次引用文献数量
0 个优先权个数
0 个技术分类数量
1 个无效请求次数
0 个分案子案个数
0 个同族专利数
0 个专利获奖情况
无保密专利的解密
否经济价值 7.0
该指标主要指示了专利技术在商品化、产业化及市场化过程中可能带来的预期利益。 专利技术只有转化成生产力才能体现其经济价值,专利技术的许可、转让、质押次数等指标均是其经济价值的表征。 因此,若您希望找到行业内的运用广泛的热点专利技术及侵权诉讼中的涉案专利,推荐您重点关注该指标。
部分指标包括:
申请人数量
1申请人类型
院校许可备案
0 次权利质押
0 次权利转移
0 个海关备案
否法律价值 19.0
该指标主要从专利权的稳定性角度评议其价值。专利权是一种垄断权,但其在法律保护的期间和范围内才有效。 专利权的存续时间、当前的法律状态可反映出其法律价值。故而,若您准备找寻权属稳定且专利权人非常重视的专利技术,推荐您关注该指标。
部分指标包括:
存活期/维持时间
6法律状态
有权-审定授权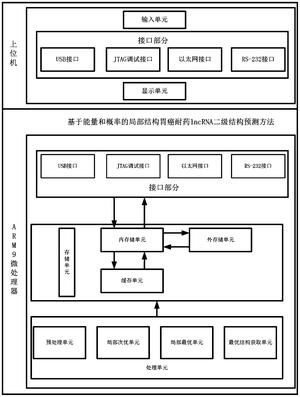











 苏公网安备 32041202001399号
苏公网安备 32041202001399号



 loading...
loading...